GenStore: A High-Performance In-Storage Processing System for Genome Sequence Analysis
作者:Nika Mansouri Ghiasi等人, lead by Onur Mutlu
@ ETH Zurich(Switzerland)
本文指出了基因序列分析的性能瓶颈为存储设备的I/O,为了解决这一问题,通过在近数据端添加两个加速器实现对数据的筛选,实现了性能的大幅提升。
基因序列分析背景知识
端到端的基因序列分析
应用场景
药物开发、病毒溯源和进化研究
主要目的
识别样本基因组和已知基因组之间的可能匹配点和差异点。
基本步骤
- 采样以及DNA样本测序:现代测序技术无法测序整个DNA,只能在这个DNA上随机采样一些短的子序列,并对他们进行测序。
- 转换:将测序到的波形图转换成碱基对(A,G,C,T等基本组成单元)的表示,这些表示被称为read(译:待匹配序列),以便存储和处理;
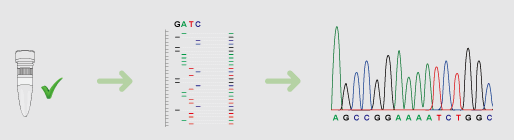
图1 DNA序列转换
- 序列匹配(Read Mapping):识别待匹配序列与参考基因组之间的匹配点和差异点,进而还原出整个DNA。
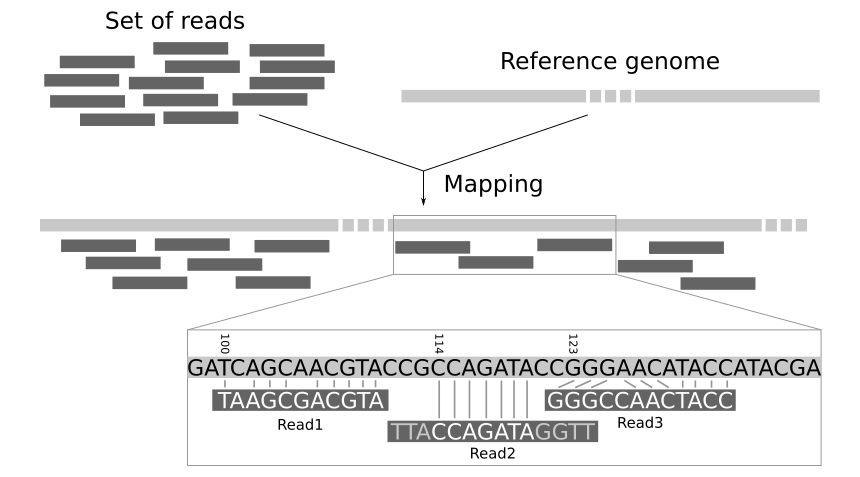
图2 序列匹配
对于同一样本,测序和转换只需要进行一次。而序列匹配出于需要与多种参考基因组进行配对、提升匹配的精度等考虑,可能要进行多次。又因为其匹配算法时间的复杂度高,成为了整个端到端基因序列匹配的瓶颈。因此本工作主要针对序列匹配进行加速优化。
序列匹配
由于参考基因组与待测序列大小不匹配,直接匹配的搜索空间太大。一般采用对参考基因组建立索引的方法来缩小搜索空间。即把整个参考基因组分成若干个长度为k的碱基序列(k-mer),然后把待测序列也分成若干个长度为k的碱基序列,对小片段进行匹配,最后通过算法拼接找到匹配位点。下图展示了怎么把一段序列分成多个5-碱基序列。

图3 产生5-碱基序列
我们用Key-Value方式记录索引结构,Value为此k-碱基序列的匹配位置。上图的一个key-value对为:<AGACC, 5>,即AGACC在5号碱基位置可以完全匹配上。
整个序列匹配过程如下
- 寻找潜在匹配位点:待测序列的k-碱基序列通过遍历查询KV表,找到所有可能匹配的k-碱基序列和位点。
- 过滤掉不可能匹配或完全匹配的待测序列
- 序列对齐:所有筛选过的位点与待测序列用计算复杂度很高的近似字符串匹配算法(ASM)计算得分,最终返回ASM达到最高分的位点,即为待测序列的目标匹配位点。
在以上计算步骤中,最耗时的是采用ASM方法进行序列对齐,是序列匹配中的一个性能瓶颈。
机会:过滤序列
由于ASM过程是处理过程中的性能瓶颈,为了最大程度地减少进行ASM的待测序列数量,过滤阶段就尤为重要。可以通过简单算法过滤掉的序列分为两种:
- 完全匹配的待测序列:待测序列可以完美与参考基因组匹配,不需要ASM来验证了;
- 无法匹配的待测序列:待测序列和参考基因组之间在匹配位点上就有很多差异,不需要ASM做匹配。
前人的大量工作主要集中在两个方向:
- 设计不同的过滤机制 → 减少ASM的调用次数
- 设计加速器或算法 → 提升ASM的性能
这个事情之前别人是怎么做的,存在什么问题?
有三种方案来提升序列匹配(read mapping)的性能:
- 设计高效的、启发式的ASM算法;
Fast Gap-affine Pairwise Alignment Using the Wavefront Algorithm【Bioinformatics, 2021】
- 主机侧的ASM硬件加速器;
Genesis: A Hardware Acceleration Framework for Genomic Data Analysis.【ISCA, 2020】
- 主机端的过滤器,剪枝掉不需要ASM的子序列。
Acceleration of Long Read Pairwise Overlapping in Genome Sequencing: A Race between FPGA and GPU.【FCCM, 2019】
以上方案都没有考虑系统的I/O开销!实际计算中,大量数据从存储系统流向主机端memory和加速器的计算单元。主机端完成的过滤器并没有从根本上减少数据在主机和存储设备之间的流动,而其中有很多数据是可以被过滤掉且不会重用的。
论文为什么要做这个事情?
为了验证采用高效的近数据处理过滤机制能够明显提升基因序列匹配的性能。文章首先通过一系列Motivational Studies来证明这一点。
评估了5个系统实现方案
- Base:主机侧SOTA软件Minimap2
- SW-filter:主机侧SOTA软件Minimap2 + 主机侧过滤器
- Ideal-ISF:主机侧SOTA软件Minimap2 + 近存储端过滤器
- ACC:主机侧SOTA硬件加速器
- Ideal-ISF + ACC:主机侧SOTA硬件加速器 + 近存储端过滤器
4种系统配置:
SSD-L,SSD-M, SSD-H代表三种SSD,分别采用SATA3、PCIe Gen3 M.2和PCIe Gen 4的I/O接口;以及DRAM:所有处理数据已经提前被load到DRAM中,无需I/O。
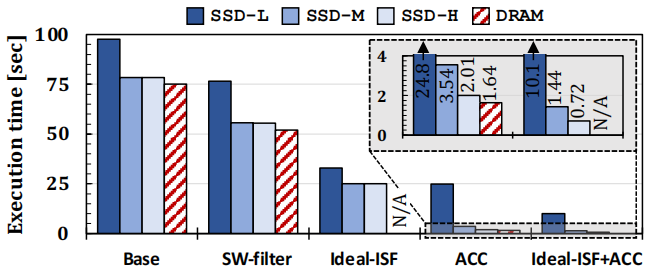
图4 Motivational Study结果
- 观察1(Ideal-ISF vs. Base & SW-filter / Ideal-ISF + ACC vs. ACC)
近数据过滤器相比于其他系统表现出了大幅度的性能提升。
分析:① 从根源上减少数据的搬运;② 减少了大概80%的无用数据的过滤,减轻了其他部分的算力负担。
- 观察2 (In Base & SW-filter, SSD-L vs SSD-H)
使用更好的SSD以提升外部带宽,能够很大程度上提升系统性能,换用DRAM反而没有很大的性能提升了。
分析:采用低端SSD时,应用是I/O瓶颈的。然而换用更好的SSD后(SSD-H或DRAM),系统的性能瓶颈又变成了CPU或主存。
- 观察3 (SW-filter DRAM vs. Base DRAM vs Ideal-ISF)
采用主机侧过滤器仅能够提升41%的性能,而采用存储侧过滤器时则体现出大幅度性能提升。
分析:在主机端使用过滤器会与主程序抢占系统资源;而在存储端添加过滤器能够减轻整个系统在过滤上的负担。
- 观察4(ACC vs. Ideal-ISF + ACC)
当主机端有加速器时,采用SSD-H比采用DRAM方案慢23%。
分析:主机端有加速器的情况下,I/O带宽重新成为了系统瓶颈。而采用Ideal-ISF + ACC的方案能够有效提升系统性能,说明在采用主机端加速器场景下,数据转移是新的系统瓶颈。因此,减少数据转移,进而加速本应用是值得一试的系统设计方案。
综上所述,在存储端做过滤器有两大好处:
- 从观察2、3可以得知,当系统带宽足够大的时候,处理性能成为了系统的瓶颈。采用主机端的过滤器会与系统抢占资源,因此,我们可以采用近存储的过滤器来减少系统的工作负担;
- 从观察4可知,当系统的计算资源充足的时候,I/O总量重新成为了系统的瓶颈。采用主机端过滤器并不会减少存储到主机的I/O总量,因此,我们可以采用近存储的过滤器来减少系统的I/O总量。
该论文解决了什么难点,难点存在的原因是什么,作者是如何发现这些难点的?
主要以下三个难点:
- 性能上:存储端的过滤器性能要高,使得近存储端的过滤能够与主机端的序列匹配在时间上重叠;
- 序列的多样性:由于测序仪器的不同,产生的序列长短、错误率也差异明显。测序仪器明显分为两种:
- 产生短序列且错误率低,匹配位点会很多;
- 产生长序列但错误率高,不太好与基因组进行匹配。
针对不同长度和不同的错误率,要求近存储过滤器能够分门别类处理。
- 硬件开销上:最好不要有明显的额外硬件开销。
针对以上难点,作者各做了哪些优化,优化背后的思想是什么?
系统概况
文章提出了第一个基因序列分析的近存储处理系统:GenStore,通过利用低开销且精确的近存储过滤器,减少了数据转移和计算开销。
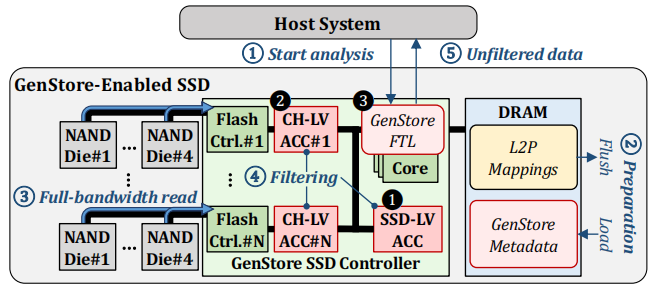
图5 GenStore整体架构图
主要的改进点:一个SSD级别的加速器;多个channel级别的加速器。用新的GenStore-FTL来管理数据流和控制流以及与主机通信。
处理流程:
① HOST发出分析指令;
② SSD进行准备:将传统的FTL数据刷新到Flash上,将GenStore必要的额外数据加载进来;
③ 从所有NAND flash芯片上读取数据,充分利用NAND flash的带宽。(与④同步)
④ 通过设计两个加速器,对不需要进一步分析的待测序列进行过滤。(与③同步)
⑤ 将没有被过滤掉的数据传送给HOST,通过ASM算法进行详细分析。
③能够实现充分利用带宽,是因为在数据预处理的时候对数据存放位置进行了仔细设计,使得每次访问都能够充分利用channel间的并行性。下面主要分析④中两个加速器的详细设计的详细设计,这两个加速器针对之前我们说的两种情况:
① 完全匹配(Exactly Matching, EM)的序列:能够与参考基因组完美匹配或者只有1~5个碱基差异的序列;
② 无法匹配(Non-Matching, NM)的序列:由于基因突变或测序时的误差,导致无法与参考基因组匹配序列。
完全匹配序列筛选加速器:GenStore-EM
针对场景
测序产生的短待测序列,有很低的错误率。根据统计,在数据集中有接近80%序列是短序列,且这部分序列能够完全匹配到目标基因组上。
设计挑战
识别每一个序列的完美匹配都需要对两个大型数据结构进行多次随机访问:
① 访问k-碱基序列索引列表,找到匹配的位置。这是一个随机的搜索;
② 访问参考基因组,找到匹配位置的完整序列,然后进行是否完美匹配的判定。
主要设计
通过仔细设计额外数据的结构和数据的形式,可以顺序化数据访问特征。文章设计了一个新的排序的k-碱基索引结构。
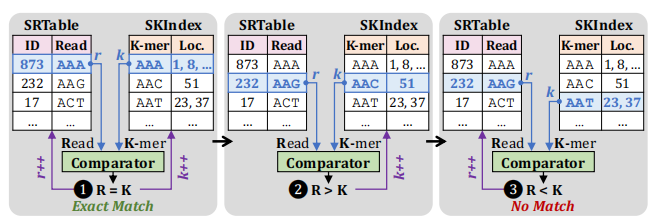
图6 排序的k-碱基索引结构
SKIndex是参考基因组的k-碱基索引结构,其中k的大小设定为了序列长度,图中假设序列长度为3。SRTable是待匹配序列表。完美匹配的流程通过流式处理两个表来实现。
主要流程如下所示:
① 若待匹配序列表中的值跟索引结构的键相同(R=K),则发现了一个完美匹配。可以直接返回结果;
② 若待匹配序列表中的值大于索引结构的键**(R>K)**,指向键值表的指针往后移;
③ 若待匹配序列表中的值小于索引结构的键**(R<K)**,说明这个序列找不到完美匹配,指向待匹配序列表的指针往后移。
完美匹配的判定可以通过采用比较器逻辑来完成,可以充分流水化筛选过程。
实现
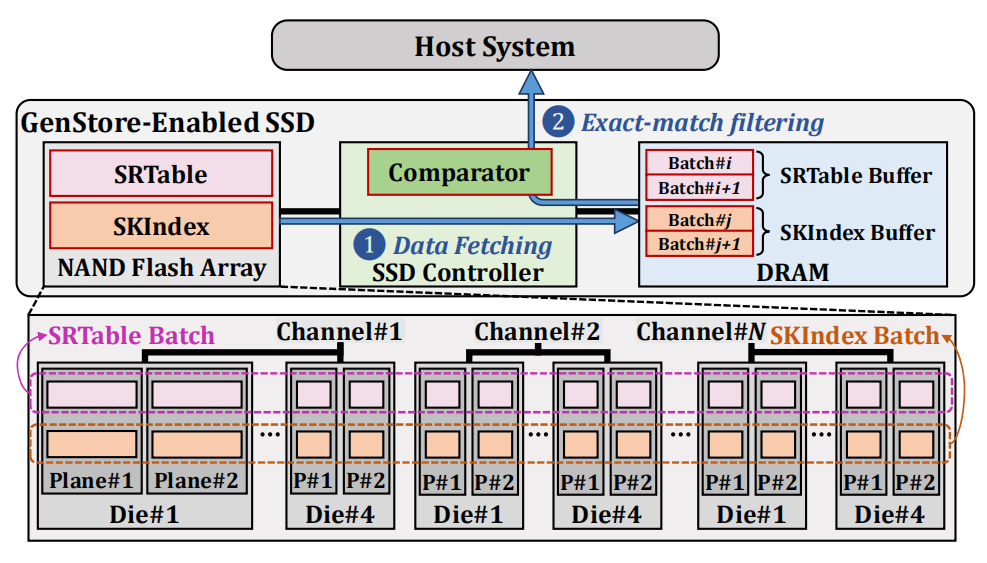
图7 GenStore-EM的实现细节
① 取数据:从NAND Flash中,以batch为单位读取SRTable和SKIndex两个数据结构;
在这一步为了充分利用了SSD的内部带宽,作者采用了两个方法。一是SRTable Batch和SKIndex Batch都是每个plane的整数倍,以保证100%的flash利用率;二是GenStore用了两个Buffer,一个处理,另一个通过flash读取数据。
② 完美匹配筛选:使用了比较器的逻辑完成完美匹配的筛选。
步骤①和②都是流水线处理的,完成后将未被筛选的数据返回给主机端。步骤①的时间要比步骤②长,通过提升存储器的内部带宽能够实现性能的进一步扩展。
无法匹配筛选加速器:GenStore-NM
针对场景
测序产生的较长的待匹配序列,错误率也比较高。以往都是采用链式分析(chaining)的方法:首先找到所有匹配位点,之后采用动态规划的算法,将重叠的匹配位点组合成长链,算法的优化目标是相似度。最终筛选掉相似度低于阈值的待测序列。
设计挑战
链式分析加速器需要执行昂贵的动态规划算法,可能会产生显著的性能开销。
主要设计
GenStore-NM设计为仅对扫描出极少量匹配位点的序列执行链式分析算法,将匹配位点多的序列直接发送到主机系统。上述设计可以由下图来验证:
图8 匹配位点与匹配成功概率的关系
图的横坐标为匹配位点数量,纵坐标为与参考基因组匹配成功的概率。可以看出,当匹配64个位点以上时,这个序列基本上能够与参考基因匹配的。因此,我们可以通过简单的匹配位点数量判断把这部分筛选工作去掉,而匹配64个位点及以上的序列占到了整个数据集的85%。
综上,通过简单的对匹配位点的数量加以限制,加速器能够减少85%的工作量,也能够保证原有工作的精度。
加速器实现
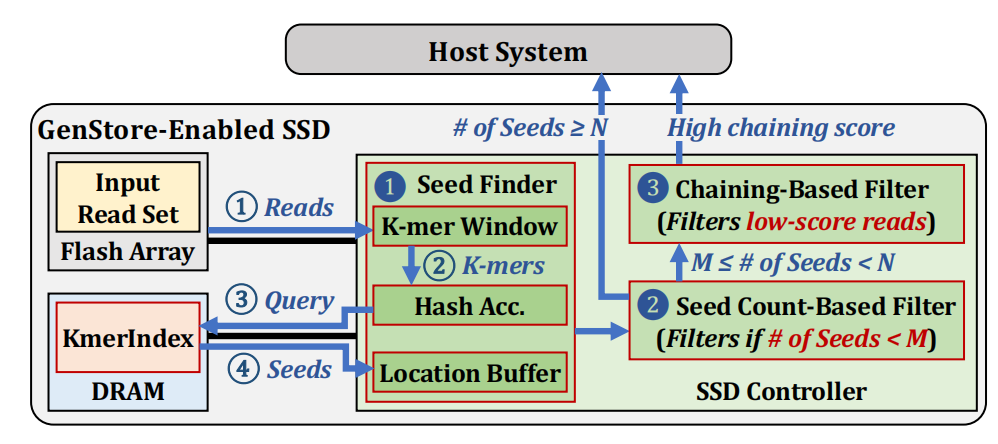
图9 无法匹配筛选加速器和系统图
① 取数据:将输入序列集合从flash上读取出来,因为这些序列比较长,因此先生成待匹配序列的k-碱基序列,这里采用滑动窗口的方式来生成。之后将待匹配序列的k-碱基序列与目标基因组的k-碱基序列相匹配,找出所有的潜在匹配位点,这里采用哈希加速器来完成。终止条件是查到了N个位点(送至HOST直接处理)或者达到了序列的尾端;所有的匹配位点被存进本地的buffer中;
② 通过匹配位点数量简单筛选:如果潜在匹配位点数量太少,以至于序列不可能匹配上,可直接筛选;如果潜在匹配位点数量太多,以至于序列大概率能匹配上,直接给HOST做处理;
③ 通过链式分析进行筛选:通过加速器设计,完成链式分析的动态规划算法,将所有相似度低的序列筛选掉,这样就完成了无法匹配序列的筛选。无法筛选的部分直接被送入HOST来完成最后ASM算法的匹配。
链式分析的动态规划公式以及对应的PE如下图所示:
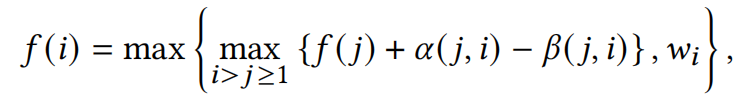
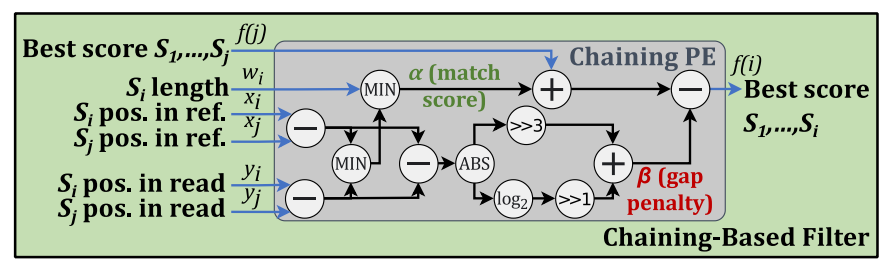
图10 链式分析加速器的PE设计
步骤①、②、③都是流水线处理的,且可以完成channel级别的加速。
实验结果
实验设定
- Base:Minimap2, GenCache(短序列匹配加速器)和Darwin(长序列匹配加速器)
- GS-Ext:Base + 存储器外部的过滤器(用与GS作对照,看I/O是否是性能瓶颈)
- GS:Base + 存储器内部的过滤器
验证平台:
采用MQ-Sim作为SSD的模拟器平台,Ramulator作为DRAM的模拟平台。软件方案是完全采用实际系统进行测试的。仍然对SSD-L, SSD-M, SSD-H三种带宽情况进行分析。
面积开销

图11 面积开销
总的面积开销非常小,小于在SATA SSD控制器中的ARM R4处理器的9.5%。
性能对比
- GenStore-EM
SIMD是在HOST端采用了以往的过滤机制的方案。
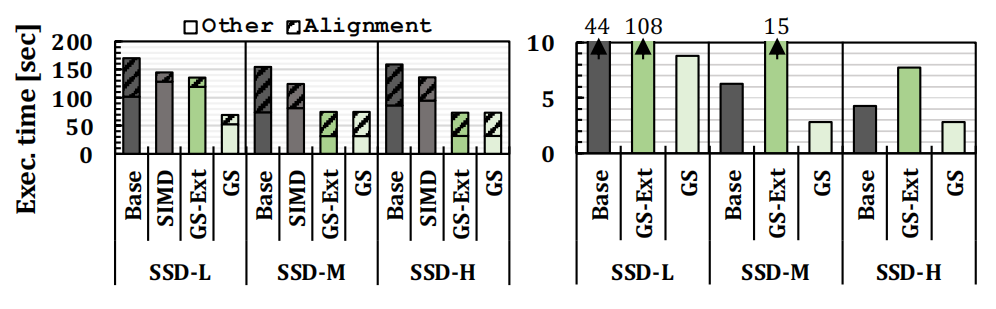
根据图(a),GS比Base快2.07-2.45倍,比SIMD快1.66-2.09倍。这是因为减少了数据搬运,且减少了主机端的工作负担。GS-ext由于I/O带宽的影响,在低端SSD上也差于GS方案,但是这一点可以通过改用更好的SSD来获得改进。SIMD不如GS-Ext方案效果好,这是因为SIMD没有优化随机访问等影响性能的因素。
图(b)是在启用了硬件加速器后的实验结果。首先,GS比Base性能提升了1.52~3.32倍。第二,GS-Ext要比Base慢很多,这是因为在存储器外的加速器需要访问很大的额外数据结构,这个访问会受到带宽的限制。
因此,采用近存储端筛选的方法能够有效解决序列匹配中的I/O瓶颈问题,尤其是当采用硬件加速器时,I/O成为了系统的新瓶颈,采用近存储的方法是进一步提升系统性能的有效手段。
- GenStore-NM
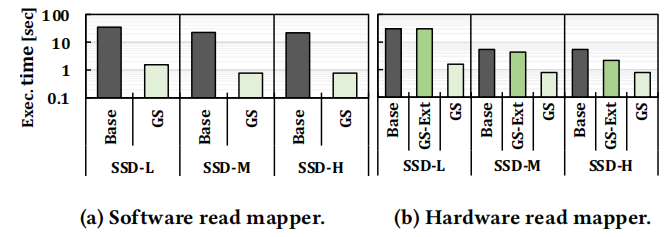
根据图a,GS比baseline性能提升22.4~29.0倍左右。其性能提升原因同上。
根据图b,GS性能提升了6.85~19.2倍,GS-ext在SSD-L和SSD-M都没有很大的性能提升,这是因为被I/O带宽限制了性能,而采用近数据的方案可以很大程度上提升系统性能。有更广泛的应用场景。
自己的思考
- 近数据处理的定位是轻量级计算,实际上比较适合进行数据的筛选;有时候可能不需要在SSD内部完成全部计算。(减少数据传输+减少计算量)
- 文章中关于I/O瓶颈还是计算瓶颈的分析值得学习,在实验对比中考虑了一个很重要的环节:加速器为什么要放在SSD上而不是HOST上,采用对比GS和GS-ext,用控制变量法来说明;
- 在计算本身不好优化的情况下,可以多考虑怎么从数据集的特征上完成剪枝。比如GenStore中根据匹配位点的数量进行初步筛选,可以将筛选的工作任务减少85%之多。这是因为作者发现了“匹配位点超过64的序列基本上都能够被匹配上,不需要做筛选”这一规律。
- 本文作者: Zhang Xinmiao
- 本文链接: https://recoderchris.github.io/2022/03/18/genstore/
- 版权声明: 本博客所有文章除特别声明外,均采用 MIT 许可协议。转载请注明出处!


